染色體結構捕捉技術
染色體結構捕捉技術 (3C, Chromosome Conformation Capture)
國立臺灣大學動物所碩士陳政儀
自從人類開始使用顯微鏡觀察細胞以來,細胞核內的絲狀染色體構造一直為科學家所好奇。隨著逐步建構的遺傳學與生物化學概念,包括:由四個鹼基構成密碼的DNA是生物體內的主要遺傳物質,影響性狀表現的基因位在染色體上,DNA是由雙股螺旋分子所構成,強化子 (enhancer) 影響啟動子 (promoter) 對其所屬基因的表現調控等。
現今生物學家普遍認為細胞核內的DNA構造,除了細胞複製時緊密堆疊的染色體結構以外,染色體序列之間的交互作用所產生的染色體構造,可能對細胞間期的基因表現產生影響。藉由染色體螢光原位雜交 (fluorescent chromosome in situ hybridization),序列之間的交互作用可以直接用顯微鏡觀察。另外使用生物化學方法剔除基因體內特定DNA片段,接著測定可能的目標基因表現,也能推測序列間是否有同位調控 (cis-regulation)。
然而,上述技術在一個研究計劃中,僅能探討一個或數個片段的可能功能,且受限於顯微鏡的解析度,對於研究擁有數百萬鹼基對的真核生物細胞基因調控模組,仍需要投入相當大的資源與時間。
在2002年,美國哈佛大學Dekker博士所領導的團隊於科學 (Science) 期刊發表染色體結構捕捉技術 (Chromosome Conformation Capture, 3C) 技術 (Dekker et al., 2002),將細胞核內DNA序列於空間上的交互作用關係以更為精確的方式解構並呈現。
他們的實驗首先使用低濃度福馬林 (formaldehyde) 處理細胞,使距離相近的絲狀染色體 (DNA) 產生鍵結 (cross-linking),再用限制酶 (restriction enzyme) 將大片段DNA切成小片段,使得鍵結的DNA序列形成X型構造(圖一),接著用接合酶 (ligase) 將X型DNA分子的兩端進行分子內接合 (intra-molecular ligation),以產生8字型或絲帶狀DNA構造。再以65℃左右高溫進行反鍵結 (reverse cross-linking),將第一步產生的相鄰DNA分子間鍵結打開,使接合的分子恢復成線狀或環狀。最後,以定量聚合酶連鎖反應 (quantitative PCR, qPCR) 放大產物及後續定序。
由於相鄰DNA序列有較高的機率於實驗的第一步中產生鍵結,使得實驗最後定量聚合酶連鎖反應中,相鄰序列有較多的反應產物。接著以定序結果反推序列於基因體上的位置 (mapping),如此便能推測引子的目標序列與基因體內的其他特定序列,在空間上具有何種程度的交互作用,也就能依此建構一段特定DNA序列的染色體在細胞核內的3D結構。
使用3C技術所得到的DNA結構圖譜 (structure topology),可用來解釋DNA結構上的改變,包括直鏈形成環形 (looping) DNA,如何調控特定基因的表現。還有,由於DNA片段在空間上的相互作用,會造成染色體構型上的改變,進而影響相關基因的調控,因此可研究強化子和啟動子如何影響目標基因的RNA轉錄。
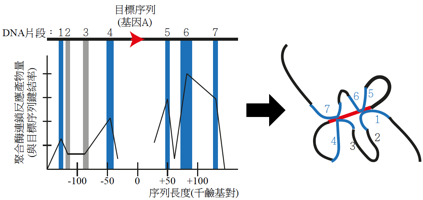
圖二 模擬3C技術所得之序列位置對照聚合酶反應產物圖與DNA構型圖譜。左圖模擬基因A經過3C技術所得之上下游序列片段(1到7)聚合酶連鎖反應產量。由於聚合酶反應產物量與相鄰DNA序列在實驗過程中形成的鍵結率成正相關,因此可以由縱軸的數值反應出片段之間在空間上的遠近,進而推測出長鏈DNA在空間上的相互作用關係圖譜。序列片段1到7中,可能部分為已知的強化子,另外功能從未被描述但與基因A有相互作用的其他片段,則可能是具有功能的強化子。因此可以藉由3C技術得到新的基因調控片段與機制。(本文作者陳政儀繪)
另外,以往功能從未被描述的DNA序列,也可因為3C技術的應用,得以讓研究人員從序列與基因片段的交互作用中,預測出新的基因表現調控機制(圖二,Jin et al., 2013)。因此,3C技術的發展為基因表現的研究帶來了相當大的突破。
然而3C技術也不是全然沒有限制,由於大部分雙倍體生物的基因只有一對序列,針對基因體中數百萬鹼基對進行放大時,稀少片段所得到的訊號往往容易被非專一性鍵結的雜訊給覆蓋。因此對照組挑選、謹慎的實驗設計、實驗數據的正確解讀(最常發生的是第一型錯誤:將實際上不存在的序列交互作用判斷為存在),有可能會造成3C實驗結果判定上的困難。另外,由於3C技術針對目標序列以聚合酶連鎖反應進行放大,在一次實驗設計中要探討整個基因體序列的交互作用必須準備相當多的引子對,仍然限制全基因體3D結構的分析 (de Wit & de Laat, 2012)。
隨著生物晶片 (microarray) 以及更便宜且高通量 (high-throughput) 的次世代定序 (next generation sequencing,NGS) 陸續發展,以3C為基礎的4C (chromosome conformation capture-on-chip,晶片染色體結構捕捉,或circular chromosome conformation capture,環狀染色體結構捕捉)、5C (chromosome conformation capture carbon copy,通用引子染色體結構捕捉) 或 HiC 等技術(圖一),被設計並應用以解構一條帶有數億鹼基對染色體的構型 (Lieberman-Aiden et al., 2009),或提升到數千個鹼基對以下的解析度(一般真核生物基因的外顯子 (exon) 大約帶有一至兩千個鹼基對),使科學家能夠更精確地預測可能的基因調控序列及染色體結構。
參考文獻
- de Wit, E., & de Laat, W. (2012). A decade of 3C technologies: insights into nuclear organization. Genes & Development, 26(1), 11-24. doi: 10.1101/gad.179804.111
- Dekker, J., Rippe, K., Dekker, M., & Kleckner, N. (2002). Capturing chromosome conformation. Science, 295(5558), 1306-1311. doi: 10.1126/science.1067799
- Jin, F., Li, Y., Dixon, J. R., Selvaraj, S., Ye, Z., Lee, A. Y., . . . Ren, B. (2013). A high-resolution map of the three-dimensional chromatin interactome in human cells. Nature, 503(7475), 290-294. doi: 10.1038/nature12644
- Lieberman-Aiden, E., van Berkum, N. L., Williams, L., Imakaev, M., Ragoczy, T., Telling, A., . . . Dekker, J. (2009). Comprehensive mapping of long-range interactions reveals folding principles of the human genome. Science, 326(5950), 289-293. doi: 10.1126/science.1181369
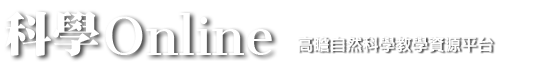

 前一篇文章
前一篇文章 下一篇文章
下一篇文章 創作搖滾樂時的大腦額葉活化
創作搖滾樂時的大腦額葉活化  治療乳癌末期新藥 FDA批准上市
治療乳癌末期新藥 FDA批准上市  伊波拉病毒(Ebola virus)—病毒的分類與特徵(上)
伊波拉病毒(Ebola virus)—病毒的分類與特徵(上)  肉毒桿菌食物中毒的機制
肉毒桿菌食物中毒的機制 ![[講座] [探索基礎科學系列講座16]命孕交響曲的四大樂章](https://highscope.ch.ntu.edu.tw/wordpress/wp-content/uploads/2016/09/Sonline.png) [講座] [探索基礎科學系列講座16]命孕交響曲的四大樂章
[講座] [探索基礎科學系列講座16]命孕交響曲的四大樂章  年齡提高卵母細胞染色體異常機率
年齡提高卵母細胞染色體異常機率  近視可能與基因有關
近視可能與基因有關  【2013諾貝爾獎特別報導】生醫獎:破解細胞傳輸系統之謎
【2013諾貝爾獎特別報導】生醫獎:破解細胞傳輸系統之謎 
