基因
[影音] CASE電影講座:侏羅紀世界─你真的要恐龍復活嗎?
[影音] CASE電影講座:侏羅紀世界─你真的要恐龍復活嗎?
◕ 時間:104年6月27日(六)19:00-21:00
◕ 地點:國立臺灣大學 思亮館國際會議廳
◕ 講者:陳俊宏 (國立臺灣大學生命科學系教授)
如果真的能親眼看到活的恐龍,就算是擠破頭,恐怕也值得!然而,不要說飼養復活的遠古動物,光是在動物園飼養像貓熊一樣稀有的動物,就需要有很多種因素的配合,除了有足夠且適當的空間及均衡的食物外,還要它們健健康康不生病,這牽涉多少知識與技術?真實動物都不見得好養?那遠古動物所需的空間及食物呢?養都不容易了,還要教育(馴養)?
再者,人類科技大幅改善人類生活,但也引發出新的問題。近年來,藉由生物技術操控生物基因,讓某些生物的演化大幅跳躍,是福?是禍?
基因與環境的互動模式
夫妻臉,其實是有科學根據的!
夫妻臉,其實是有科學根據的!
高瞻計畫特約編譯 柯廷龍/國立臺灣師範大學生命科學系 李冠群副教授責任編輯
編譯來源:People more likely to choose a spouse with similar DNA, finds CU-Boulder study
根據以往的研究發現,人們往往傾向與自己具有相同性格、教育背景、經濟狀況、年齡甚或同樣宗教理念的另一半共結連理。在臺灣,不時也常能夠聽到人說某對夫婦有「夫妻臉」。現在科學研究證實了此說法並非無稽之談。
在美國的科羅拉多大學博爾德分校(Corolado- Boulder),學者們針對825對非西班牙裔美國白種人做了一項耐人尋味的研究,研究由在博爾德分校任職的研究助理班傑明‧多名格(Benjamin Domingue)、助理教授傑森‧柏德曼(Jason Boardman)和其他同事們所執行。
專家們比對了每一對夫妻的基因以及隨機配對男女的基因,檢視了170萬個單核苷酸多型性(Single-nucleotide polymorphism, SNP)1,研究人員發現,有較多的夫婦擁有相似的基因,
細胞的命運決定機制
細胞的命運決定機制
國立臺灣大學生命科學系助教范姜文榮編譯
編譯來源:独立行政法人理化学研究所(RIKEN)2013年10月1日訊 <細胞の運命を左右する新しい分子メカニズムの一端を解明>
原文出自日本行政法人理化学研究所
從未分化的幹細胞到各類細胞的命運決定,與遺傳基因表現的啟動與關閉密切相關。幹細胞為了分化功能須維持其全能分化性1,在維持分化相關基因活化的同時,實際上也有必要能停留於非活化狀態。
為了維持幹細胞此特性,扮演重要角色的是「基團蛋白質群」2 。過去研究已指出「基團蛋白質群」能在細胞分化、增值或抑制有關基因的所在位置,形成「基團蛋白抑制複合體」3,將該基因予以不活化,因此「基團蛋白抑制複合體」是幹細胞命運決定的主要因子,但其抑制基因表現、及抑制解除的分子機制仍幾乎未知。
日本理化學研究所的研究團隊使用老鼠基因改變技術及細胞影像技術,企圖解析「基團蛋白抑制複合體」對基因調控的機制。首先製作能使用螢光追蹤「基團蛋白質群」的基改老鼠,以瞭解此複合體在細胞核內位置。再透過顯微鏡觀察基改老鼠的胚胎細胞,發現在非活化狀態的基因區域,有「基團蛋白抑制複合體」頭尾聚合、連結在一起,形成斑點狀的「基團蛋白結構體」。
為瞭解「基團蛋白結構體」的角色,有必要觀察「基團蛋白結構體」形成情形。研究團隊推論「基團蛋白抑制複合體」的結構成分之一「Phc2蛋白質」具有自我聚合活性是關鍵所在。因此製作會引發聚合形成不全的基因異常老鼠,即Phc2基因點突變老鼠,並觀察其細胞狀態。結果如同預料,「基團蛋白結構體」未形成,且與基因表現啟動或關閉有密切相關的染色質結構也同時鬆開,而受到「基團蛋白抑制複合體」所抑制的基因群表現則上升。例如Hox基因表現增加,影響脊椎骨的形成方式;Cdkn2a基因表現增加,提早引發細胞老化。檢視Phc2基因點突變老鼠,發現「基團蛋白結構體」不全,誘導原應變為頸椎的脊椎骨位置,反而變為胸椎;原應為胸椎的位置,變為腰椎,出現體節位置變換現象。以上結果顯示「基團蛋白結構體」的形成能抑制細胞命運決定的相關基因。
阻止珊瑚滅絕─解析珊瑚共生藻基因體
阻止珊瑚滅絕─解析珊瑚共生藻基因體
國立臺灣大學生命科學系助教范姜文榮
編譯來源:《Draft Assembly of the Symbiodinium minutum Nuclear Genome》
日本沖繩科學技術研究所、日本國立遺傳學研究所、日本國立生物資訊學研究所及東京大學醫學基因科學系等研究團隊,首次成功解析出與珊瑚共生的蟲黃藻1基因體,該研究團隊先前已於2011年率先成功解析珊瑚基因體,因此獲得宿主─珊瑚與共生者─蟲黃藻兩者的基因體資訊。透過基因體的遺傳資訊,將對進一步解析珊瑚與蟲黃藻間的共生關係有重要的幫助。
珊瑚礁與熱帶雨林並列為生物多樣性最豐富的地區,另因與漁業及觀光有關聯,使珊瑚礁也成為經濟上非常重要的場所。但是隨著地球的溫暖化、海水酸化等因素,使珊瑚礁面臨危機,已有研究報告指出全部地球的1/3珊瑚礁已漸漸消失,許多國家的政府單位全力進行防止地球暖化與珊瑚礁保護的工作。
為了保護珊瑚礁,有必要理解製造珊瑚礁的主角─珊瑚的生物特性,造礁珊瑚雖與水母、海葵等同樣是刺絲胞動物,但是造礁珊瑚具備其他刺絲胞動物少有的2個重要特徵:其一是造礁珊瑚與屬於渦鞭毛藻的蟲黃藻維持細胞內共生關係,因此可自蟲黃藻獲取營養物質而存活。另一特徵是具有利用二氧化碳製造碳酸鈣骨骼、形成珊瑚礁的能力。造礁珊瑚與蟲黃藻之間具有絕對的共生關係,如果發生蟲黃藻離開珊瑚或在珊瑚內死亡的情況,就會造成珊瑚白化現象,狀況嚴重時會影響珊瑚生存甚至死亡,因此有必要理解下列問題:珊瑚與蟲黃藻是如何維持其共生關係?為何海水溫上升等刺激會造成共生關係的崩解?
原致癌基因(Proto-oncogene)
原致癌基因(Proto-oncogene)
國立臺灣師範大學生命科學系研究助理胡琬琳
 原致癌基因(proto-oncogene)為細胞內之正常基因,其表現的蛋白質產物能促進細胞生長及分裂,當原致癌基因在諸如放射性物質、化學物質及病毒的作用影響下而突變(mutation)時,會造成其蛋白質產物過量產生或是活性增加,使細胞的生長及分裂大幅增加而導致癌症(cancer)。此時突變後的原致癌基因即稱為致癌基因(oncogene),換言之,原致癌基因是致癌基因的前身。
原致癌基因(proto-oncogene)為細胞內之正常基因,其表現的蛋白質產物能促進細胞生長及分裂,當原致癌基因在諸如放射性物質、化學物質及病毒的作用影響下而突變(mutation)時,會造成其蛋白質產物過量產生或是活性增加,使細胞的生長及分裂大幅增加而導致癌症(cancer)。此時突變後的原致癌基因即稱為致癌基因(oncogene),換言之,原致癌基因是致癌基因的前身。
原致癌基因突變為致癌基因的方式可分為三類:(1) 基因於基因組中(genome)的移動;(2) 原致癌基因的放大(amplification);(3) 原致癌基因本身或其調控區(control element)發生點突變(point mutation)。在第一類情形中,原致癌基因經轉位作用(translocation)移至不同的基因座(locus),因受到較為活化的啟動子(promoter)調控,導致其表現量增加而轉變為致癌基因;在第二類情形中,因為原致癌基因的套數(copy number)增加,導致其表現量增加而轉變為致癌基因;在第三類情形中,因為原致癌基因本身發生點突變,使其所表現的蛋白質活性增加或不易被降解(degradation),或者當原致癌基因調控區發生點突變,亦可導致其表現量增加而轉變為致癌基因。
目前研究所發現的原致癌基因大致可分為生長因子(growth factors)、受體酪氨酸激酶(receptor tyrosine kinases)、膜上非受體酪氨酸激酶(membrane mssociated non-neceptor tyrosine kinases)、G蛋白偶聯受體(G-protein coupled receptors)、絲胺酸/蘇胺酸激酶(serine/threonine kinases)、核內DNA結合/轉錄因子(nuclear DNA-binding/transcription factors)等基因。
以下分項舉例並簡介各原致癌基因:
1. 生長因子(growth factors)
SIS基因 [v-sis基因為猿猴肉瘤病毒(simian sarcoma virus)中的致癌基因] 表現血小板衍生性生長因子B鏈(PDGF B chain),此基因為最先被確認與已知細胞內基因具有同源性的致癌基因。
int-2基因表現一種與纖維母細胞生長因子相關的生長因子(FGF-related growth factor)。
KGF基因又稱HST基因,亦表現一種與纖維母細胞生長因子相關的生長因子,而且在胃癌及卡波西氏肉瘤(Kaposi’s sarcoma)細胞中被發現。
2. 受體酪氨酸激酶(receptor tyrosine kinases)
FMS基因表現血球生長激素-1 受體(colony stimulating factor-1 receptor),且被確認為一反轉錄病毒(retrovirus)的致癌基因
TRK基因表現類神經生長因子受體蛋白(NGF receptor-like protein),第一個被發現的TRK基因位於胰臟癌細胞中。
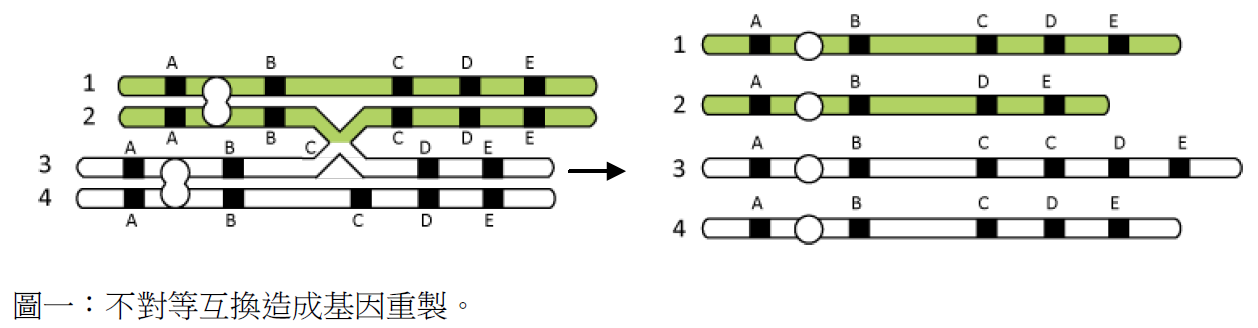
基因重製(Gene duplication)
基因重製(Gene duplication)
國立臺灣師範大學生命科學系研究助理鄭杏倩
DNA的數量(copy數)可藉由基因重製(gene duplication)事件而增加。基因重製依照參與重製之基因體長度之不同而分為以下幾個類型。
(1) 部分基因重製(partial/internal gene duplication)
(2) 完整基因重製(complete gene duplication)
(3) 部分染色體重製(partial chromosomal duplication)
(4) 完整染色體重製(complete chromosomal duplication)
(5) 多倍體化/基因體重製(polyploidy/genome duplication)
前四類型皆為區域性的基因重製(regional duplication)事件,重製後並不會改變生物體內染色體的數量。因此,一般認為多倍體化事件對演化所扮演的角色遠遠大於區域性之基因重製。然而近來的研究顯示,區域性的基因重製在演化上也扮演著舉足輕重的角色。
區域性的基因重製之發生機制如下。在細胞進行有絲分裂,同源染色體配對進行基因重組(recombination)時,發生非同源區域互換,亦即不對等互換(unequal crossing-over),使得有些配子體(gamete)帶有兩套相同的基因,而相對的有些配子體的某基因就消失了。以下用簡單的示意圖(圖一)來描述不對等互換造成基因重製之事件。
不對等互換事件常發生在染色體中有許多重複序列的地方,且一旦發生了第一次不對等互換,此區域再發生不對等互換的機率則增加(因為有更多的重複序列提供不對等互換發生),因此常常可以看到基因體中某一區域有基因家族(gene family)的狀況。當重製事件產生後,個體內的第一套基因負責正常功能運作,另外多餘的第二套基因則可以累積變異而不會影響生物之正常運作,經過長時間的天擇(natural selection)或是中性的遺傳漂變(genetic drift)後,第二套基因有機會可以累積足夠的變異而演化成具有新功能之新基因(new gene)。
基因裡的人類遠古故事
基因裡的人類遠古故事
知識通訊評論第120期
利用分子鐘技術,調和了過去基因與化石證據對遠古歷史關鍵時間的分歧,然而這個更慢的演化時間,也造成不同物種演化分歧上的明顯問題,使問題更形複雜。
以往我們只能倚靠人類祖先遺留的骨骸及工具追想他們的昔日生活,一九六○年代發現的DNA賦予我們另一種解讀方法。其中某些結果很有啟發性,例如DNA研究顯示,所有現代人都源自十萬年前非洲的祖先。但此方法所提出人類進化史關鍵時刻發生點,卻與考古學完全不符,令人費解。
多賴DNA變異速率預測的進步,可以用分子鐘來確立遺傳學年代,使得現今考古學家及遺傳學家才開始能把故事拼在一起。英國伯明罕大學的考古學家羅斯(Jeff Rose)說,「遺傳學及考古學終於能重新取得某種程度的共識,委實令人意外。」參考彼此的數據,考古學者及遺傳學者現在可以更具信心地著手處理人類歷史中種種細微問題。英國新克斯頓的衛康信託基金會桑格研究所演化基因組學家史卡利(Aylwyn Scally)說,「他們得相信這個故事是真的。」
DNA鐘的觀念很簡單:比較兩個物種基因序列上鹼基的差異,就可以推演出他們共同祖先生存的時代,距今有多久。然而這個預測成功的前提是,遺傳學家必須知道一個重要的資訊:DNA鹼基發生改變的速率。
遺傳學家早先是以人類與其他靈長類動物基因組的差異,來預測突變速率。諷刺的是,取得物種差異的時間點的方法,卻是靠化石證據,而以此為基礎科學家做出結論;人類的的DNA中每個鹼基發生突變的機率約為每十億年一次。德國馬克斯普朗克研究院演化人類學分子人類學家維吉蘭特(Linda Vigilant)認為,「這是一個可疑的概略數。」結果他的懷疑確實有理。
在過去幾年,藉由數十個家庭的完整基因組定序,並比較雙親與孩子的突變率,遺傳學家能夠親眼目睹分子鐘的運作。史卡利聲稱,這些研究顯示,這個鐘走的速度或許是先前預測的一半。
在發表於九月十一日的評論文中,史卡利與他的同事杜賓(Richard Durbin)採用較慢的速率,重新評估人類演化中的關鍵時間分歧點。「如果突變的速率降低為一半,所有預估的年代都會增為兩倍。」史卡利說,「這樣會產生南轅北轍的結果。」然而最近分子鐘預估的年代,與考古學上的關鍵時程更加吻合。
舉例來說,西班牙阿塔波卡地區的胡瑟裂谷出土的四十萬至六十萬年前海德堡人骨,是尼安德特人的直接祖先。而基因研究推測,尼安德特人的先祖,是在更為近期約二十七萬至四十三萬五千年前,由演化成現代人的一個分支分化出去的。比較慢的分子鐘,將此一分支日期推到更為可靠的六十萬年前。


![[影音] CASE電影講座:侏羅紀世界─你真的要恐龍復活嗎?](https://highscope.ch.ntu.edu.tw/wordpress/wp-content/uploads/2015/07/ap1.png)